Data Science beschleunigt die Welt.
Wir synchronisieren Ihr Unternehmen.
Kontakt
Newsletter abonnieren
„*“ zeigt erforderliche Felder an
Die meisten kommen mit R als erstes über das mächtige RStudio in Kontakt. Damit fällt das Schreiben von einfachen Analyse-Skripten leicht. Doch für die Nutzung von Analysen oder Modellen aus R in Produktivsystemen ist RStudio ungeeignet. Hier zeigen wir Ihnen, wie Sie mit wenig Aufwand aus Ihrem R-Skript ein Helferlein für die Kommandozeile machen.
Zusammenfassung:
Wenn Sie Analysen mit R machst, werden Sie sehr wahrscheinlich regelmäßig RStudio verwenden. Zwar gibt es auch Alternativen, aber unter Data Scientists ist das eigentlich das Standard-Tool, um Analysen durchzuführen und Modelle zu entwickeln.
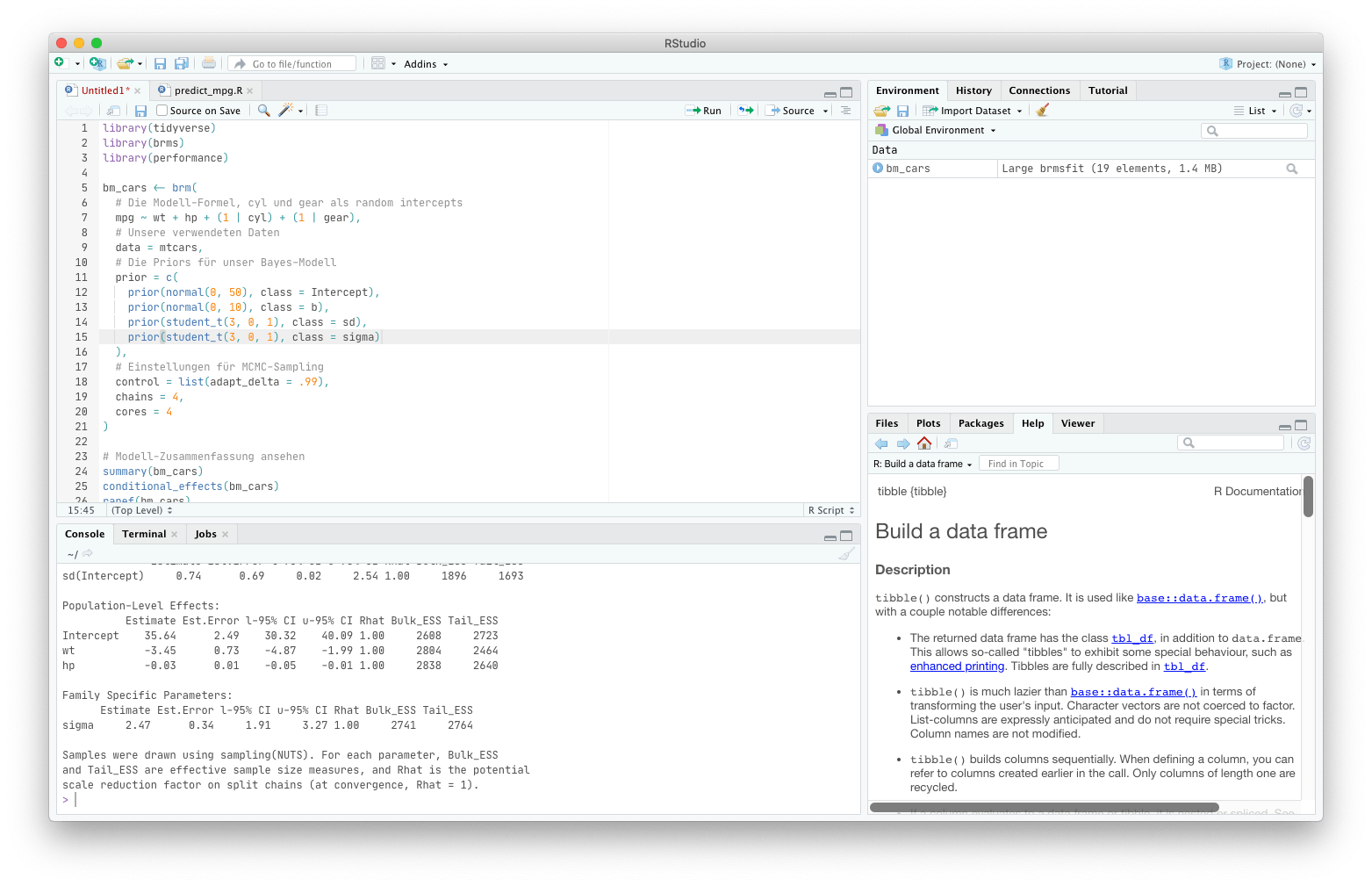
Abbildung 1: So sieht es aus, wenn wir Analysen in RStudio entwickeln.
Was Sie in RStudio im Code-Editor (im Screenshot oben links) schreiben und abspeichern, ist für sich bereits ein R-Skript. Also eine Datei, in der Ihre gesamte Analyse geschrieben steht. In der Praxis werden Sie wahrscheinlich häufig immer wieder einzelne Abschnitte mittels „Run“ starten. In den meisten anderen Programmiersprachen hingegen werden solche Skripte einfach von oben nach unten „durchgearbeitet“ und Zeile für Zeile ausgeführt – in etwa so wie das, was R macht, wenn Sie den „Source“-Button verwenden.
Nun können Sie ein geschriebenes R-Skript aber auch ähnlich wie mit Python komplett durchlaufen lassen, so dass R jede Zeile „ausführt“, ohne dass Sie das Skript in RStudio geöffnet haben. Genau dafür ist dann die Kommandozeile praktisch. Wenn Sie dort im gleichen Ordner wie Ihr Skript sind und
Ein R-Skript über die Kommandozeile ausführen:
Rscript my_script.Rin der Kommandozeile eingeben, wird Ihr R-Skript von oben nach unten durchgearbeitet. Und auf diese Weise stehen Ihnen für die Implementierung Ihres Machine-Learning-Modells viele Einsatzmöglichkeiten zur Verfügung.
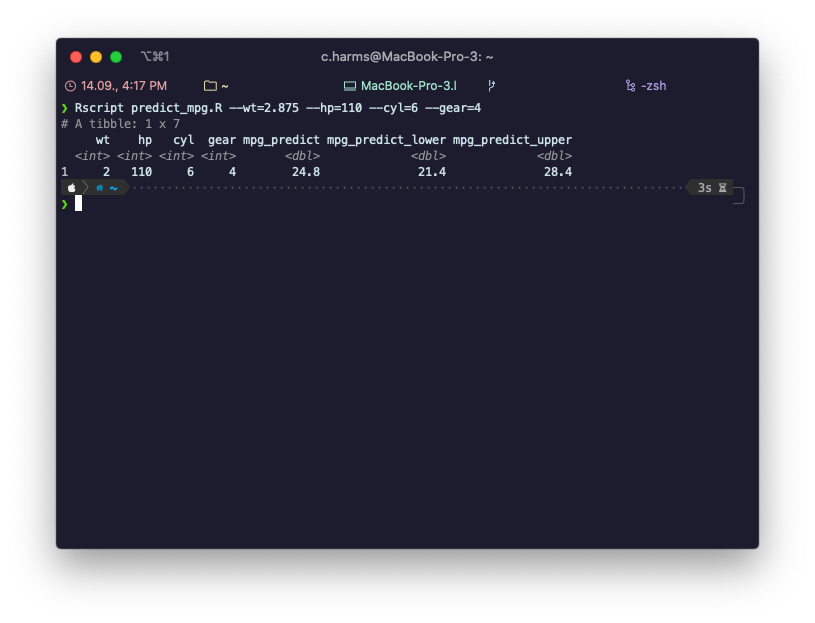
Abbildung 2: So kann das Terminal (oder die Kommandozeile) aussehen, wenn man ein R-Skript zur Vorhersage von Daten verwendet.
Vielleicht fragen Sie sich, was genau die Kommandozeile (auch Terminal genannt) ist. Je nachdem, welches Betriebssystem Sie nutzen, kommen Sie auf unterschiedliche Weisen mit der Kommandozeile in Berührung. Als Linux-Nutzer werden Sie es gewohnt sein, über die so genannte Shell zu arbeiten. Unter Windows („Kommandozeile“ oder „command line“) oder macOS („Terminal“ oder „zsh“) ist das seltener notwendig, aber eben auch möglich. So oder so: Über das Terminal können Sie mittels Texteingabe viele System-Befehle nutzen, Programme starten und Dich durch das Dateisystem bewegen. Sobald Sie sich von Ihrem Desktop-Rechner in die Cloud bewegen, ist das Terminal oft die einzige Möglichkeit, Programme auf einem Server zu starten. Hier gibt es meistens keine grafische Oberfläche, wo Sie RStudio nutzen könnten. Kurz gesagt: Sobald Sie R-Skripte unabhängig von Ihrem eigenen Computer benutzen möchten, sollten Sie sich die Frage stellen, wie Sie Ihre Analysen für die Arbeit mit der Kommandozeile vorbereiten.
Bei uns geht es häufig darum, Prozesse in denen Machine-Learning-Modelle zum Einsatz kommen, zu automatisieren. Dabei wollen wir nicht immer von Hand das dazugehörige R-Skript in RStudio durchlaufen lassen, sondern wie oben beschrieben das Skript als kleines Helferlein benutzen, das über die Kommandozeile ausgeführt werden kann. Wenn es darum geht, Analysen und Modelle im produktiven Einsatz zu haben, stellt sich meist die Frage nach Schnittstellen. Über solche Programmierschnittstellen, auch API (Application Programming Interface) genannt, können Daten analysiert und das Ergebnis anschließend abrufbar gemacht werden. Wenn es eine Vielzahl von solchen kleinen Tools gibt, die einen bestimmten Zweck erfüllen, spricht man auch von Microservices, die häufig auch auf virtuellen Computern in der Cloud laufen.
Um unsere eigenen Modelle für Vorhersagen und Textanalysen für uns und unsere Kunden nutzbar zu machen, haben wir eine Infrastruktur für solche Microservices entwickelt (getauft auf den Namen Carbon). Jedes unserer Modelle ist dann ein kleines Kommandozeilen-Tool, das über eine entsprechende API aufgerufen werden kann – egal, von welchem Rechner in unserem Netzwerk und weitestgehend unabhängig davon, ob die Ergebnisse dann in R, Python oder Alteryx weiterverwendet werden.
Anhand eines sehr einfachen Modells wollen wir Ihnen nun zeigen, wie man einfache Kommandozeilen-Tools mit R schreiben kann, die sich dann flexibel einsetzen lassen.
Natürlich bedienen wir uns für dieses Beispiel eines recht einfachen Datensatzes, den wir in R als Beispieldatensatz direkt verfügbar haben: mtcars. In diesem Datensatz sind mehrere Fahrzeuge mit ihren Eigenschaften enthalten. In unserem kleinen Modell wollen wir den Verbrauch (Spalte mpg) auf Basis der Leistung (Spalte hp), der Anzahl der Zylinder (Spalte cyl) und der Anzahl der Gänge (Spalte gear) vorhersagen. Da uns die Unsicherheit in unserer Vorhersage interessiert, nutzen wir ein Bayes’sches Mixed Effects Modell, das wir mit brms berechnen werden. Am Ende haben wir ein trainiertes Vorhersagemodell (predictive model), mit dem wir auf Basis von Gewicht, Pferdestärken, Anzahl der Zylinder und Anzahl der Gänge den Spritverbrauch von Fahrzeugen vorhersagen können.
Das bisherige Skript, in dem wir unser Modell in RStudio entwickelt haben, sieht vielleicht so aus:
Das Modell-Training in R:
library(tidyverse)
library(brms)
library(performance)
bm_cars <- brm(
# Die Modell-Formel, cyl und gear als random intercepts
mpg ~ wt + hp + (1 | cyl) + (1 | gear),
# Unsere verwendeten Daten
data = mtcars,
# Die Priors für unser Bayes-Modell
prior = c(
prior(normal(0, 50), class = Intercept),
prior(normal(0, 10), class = b),
prior(student_t(3, 0, 1), class = sd),
prior(student_t(3, 0, 1), class = sigma)
),
# Einstellungen für das MCMC-Sampling
control = list(adapt_delta = .99),
chains = 4,
cores = 4
)
# Modell-Zusammenfassung ansehen
summary(bm_cars)
conditional_effects(bm_cars)
ranef(bm_cars)
# Posterior Predictive Checks
pp_check(bm_cars, type = "dens_overlay")
# Vorhersage-Fehler
model_performance(bm_cars)
# Modell für spätere Nutzung bei der Vorhersage
saveRDS(bm_cars, file = "./bm_cars.Rds")Wenn Sie bisher noch keine Erfahrung mit brms gemacht haben: Kein Problem. Im Wesentlichen bauen wir hier eine einfache lineare Regression. Zwei Dinge unterscheiden sich zum klassischen Ansatz mit lm: Wir haben Priors, eine Eigenschaft von Bayes-Modellen, und wir betrachten die Anzahl Zylinder und die Anzahl Gänge als Faktoren, die je Ausprägung eine eigene Konstante in der Modellformel haben. Wenn Sie mehr darüber erfahren wollen: Eine Einführung zu diesen Methoden finden Sie zum Beispiel in diesem Vortrag. Mehrebenen-Modelle, oder auch Mixed-Effects-Modelle, werden wir in einem der nächsten Blog-Beiträge vertiefen, aber diese Seite von Michael Freeman stellt die Grundidee sehr anschaulich dar.
In der Praxis würden wir natürlich die Modellentwicklung systematischer angehen: Verschiedene Modelle mittels Fehlerstatistiken oder Information Criteria vergleichen, Kreuzvalidierung zur Absicherung unserer Vorhersagegenauigkeit nutzen und weitere Prädiktoren aufnehmen, um die Genauigkeit zu verbessern. In diesem Fall soll aber zunächst dieses einfache Beispiel genügen.
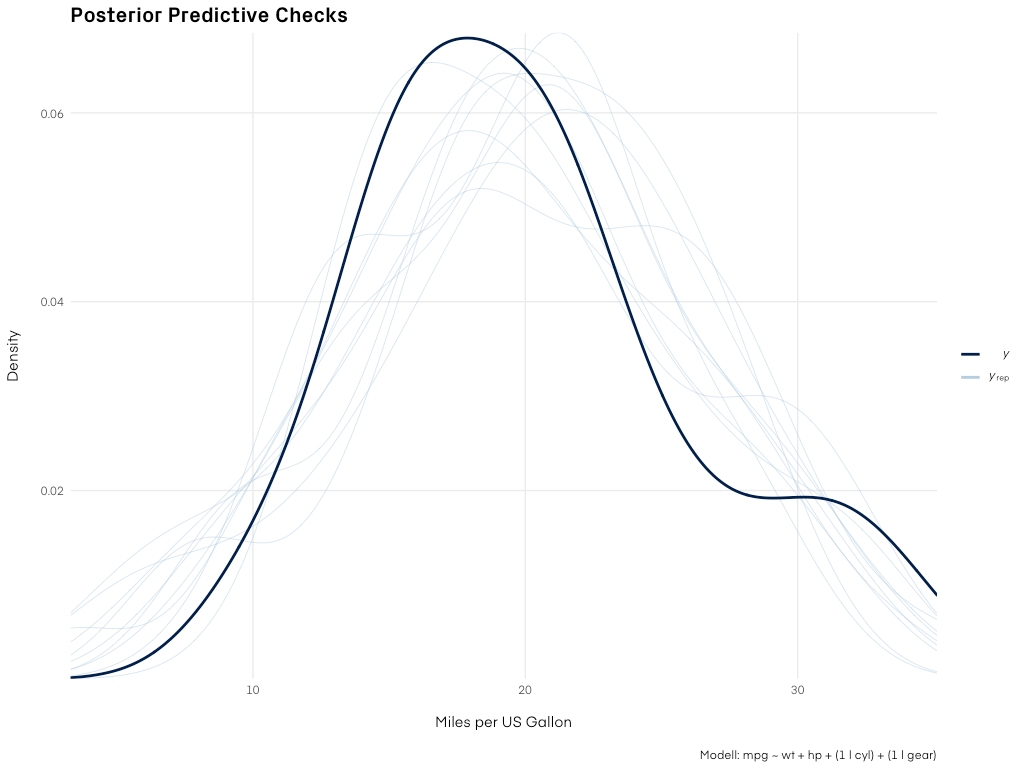
Posterior Predictive Checks
Abbildung 3: Die Vorhersagen unseres Modells verglichen mit den Daten. Für eine echte, praktische Anwendung würden wir das Modell noch weiter verbessern.
Am Ende unseres Skripts haben wir das Modell abgespeichert, das wir für neue Datenpunkte nutzen wollen, um den Spritverbrauch vorherzusagen. Für diese Vorhersage schreiben wir uns ein neues R-Skript, das wir flexibel einsetzen können.
Die wahre Macht von Kommandozeilen-Tools („command line utilities“) wie unserem R-Skript kommt durch Parameter, d. h. Informationen, die wir beim Aufruf des Skripts mitgeben und auf die unser Skript flexibel reagieren kann. Diese Parameter werden einfach an den Aufruf angehängt, zum Beispiel:
Ein R-Skript mit Parametern in der Kommandozeile ausführen:
Rscript predict_mpg.R --wt=2.62 --hp=110 --cyl=6 --gear=4In unserem Fall wollen wir alle Prädiktoren für unser Modell über Kommandozeilen-Parameter angeben, sodass wir die Informationen zu unserem neuen Auto – für das wir eine Vorhersage machen wollen – nicht in das R-Skript fest reinschreiben müssen.
In der letzten Zeile unseres bisherigen R-Skripts haben wir das Modell als .Rds-Datei in das aktuelle Arbeitsverzeichnis abgespeichert. Wenn Sie unsicher sind, wo das ist: Mit getwd() wird Ihnen das in R angezeigt. Das .Rds-Format ist ein Standard von R, um Objekte (wie unser Modell) zu speichern und später wieder laden zu können. Sehr praktisch, denn wir wollen nicht bei jeder Vorhersage das Modell aufs Neue .
Nun müssen wir in unserem neuen R-Skript also das Modell laden und die Informationen aus den Kommandozeilen-Parametern extrahieren. Es gibt verschiedene Pakete, um das zu tun. Aber das Paket optparse hat sich bei uns als das einfachste und flexibelste erwiesen. Am besten installieren Sie es direkt in R oder RStudio mit:
Paket optparse installieren:
install.packages('optparse')Das Paket liefert einige Funktionen, um die Kommandozeilen-Parameter auszuwerten. Das Herzstück ist dabei die Funktion make_option, die wie folgt verwendet werden kann:
Einen Kommandozeilenparameter hinzufügen:
make_option(c("--wt"), # Wie sieht der Parameter in der Kommandozeile aus?
type = "integer", # Um was für ein Datentyp handelt es sich?
dest = "wt", # Wie heißt die Variable, in der der Parameter-Wert gespeichert werden soll?
default = NA, # Der Standardwert, wenn der Parameter nicht angegeben wird
metavar = "WEIGHT", # Ein Platzhalter-Wert für die Hilfe (siehe unten)
action = "store", # Was soll mit dem Parameter passieren? "store" bedeutet, dass der Wert in einer Variablen gespeichert wird
help = "The car's weight (in 1000 lbs)."
# Ein kurzer Text, der den Parameter beschreibt
)Diesen Block müssen wir für jeden Parameter, den wir nutzen möchten, wiederholen und dann über zwei weitere Funktionen zusammensetzen. Am Ende sieht der gesamte Block so aus:
Automatische Hilfe in der Kommandozeile:
suppressPackageStartupMessages(library(optparse))
cli_options <- list(
make_option(c("--wt"), type = "integer", dest = "wt",
default = NA, metavar = "WEIGHT", action = "store",
help = "The car's weight (in 1000 lbs)."),
make_option(c("--hp"), type = "integer", dest = "hp",
default = NA, metavar = "HORSEPOWER", action = "store",
help = "The car's gross horsepower."),
make_option(c("--cyl"), type = "integer", dest = "cyl",
default = NA, metavar = "CYLINDERS", action = "store",
help = "Number of cylinders."),
make_option(c("--gear"), type = "integer", dest = "gear",
default = NA, metavar = "GEARS", action = "store",
help = "Number of forward gears.")
)
args <- parse_args(OptionParser(option_list = cli_options))Wenn wir nun dieses R-Skript als predict_mpg.R abspeichern und mit Rscript über die Kommandozeile aufrufen, passiert erstmal nicht sehr viel – aber optparse legt automatisch den Parameter -h an, mit dem es eine kurze Hilfe gibt:
Alle benötigten Parameter hinzufügen:
❯ Rscript predict_mpg.R -h
Usage: predict_mpg.R [options]
Options:
--wt=WEIGHT
The car's weight (in 1000 lbs).
--hp=HORSEPOWER
The car's gross horsepower.
--cyl=CYLINDERS
Number of cylinders.
--gear=GEARS
Number of forward gears.
-h, --help
Show this help message and exitDas ist sehr praktisch, damit andere Nutzer wissen, was sie mit diesem Skript machen können und es orientiert sich an den Konventionen für Kommandozeilen-Programme aus der Linux-Welt.
Bevor wir zur eigentlichen Logik kommen, sollten wir noch sicherstellen, dass der Nutzer unseres kleinen Skripts alle wichtigen Parameter angegeben hat. optparse speichert alle Werte in einer Liste, die wir über die oben zugewiesene Variable args abfragen können. So können wir sehr einfach alle Parameter überprüfen und bei Bedarf das Skript mit quit() abbrechen:
Parameter-Angaben überprüfen:
suppressPackageStartupMessages(library(cli))
# Angegebene Parameter überprüfen
if (sum(is.na(args)) > 0) {
cli_alert_danger("Alle Angaben zum Fahrzeug müssen angegeben werden.")
quit()
}Das Paket cli, das bei R immer dabei ist, stellt einige praktische Funktionen bereit, mit denen man Warnhinweise schön aufbereitet darstellen kann. Das sieht dann zum Beispiel so aus:
Wenn die Prüfung fehlschlägt:
❯ Rscript predict_mpg.R
x Alle Angaben zum Fahrzeug müssen angegeben werden.Die eigentliche Logik des Skripts besteht aus:
Im Skript sieht das dann so aus:
Vorhersagen mittels unseres Modells generieren:
suppressPackageStartupMessages(library(tidyverse))
suppressPackageStartupMessages(library(brms))
new_df <- tibble(wt = args$wt,
hp = args$hp,
cyl = args$cyl,
gear = args$gear)
bm_cars <- readRDS("bm_cars.Rds")
df_predictions <- predictive_interval(bm_cars, newdata = new_df, prob = .8)
new_df$pred <- round(predict(bm_cars, newdata = new_df)[,1])
new_df$pred_lower <- round(df_predictions[,1])
new_df$pred_upper <- round(df_predictions[,2])
print(new_df)Wenn wir nun alle Teile zusammenwerfen und im Skript speichern (das fertige Skript finden Sie auch hier), können wir für unsere neueste Ergänzung im Fuhrpark, ein Mazda RX4 Wagon, den Spritverbrauch mit unserem Tool vorhersagen:
Das fertige Skript in der Kommandozeile mit Vorhersagen:
❯ Rscript predict_mpg.R --wt=2.875 --hp=110 --cyl=6 --gear=4
# A tibble: 1 x 7
wt hp cyl gear mpg_predict mpg_predict_lower mpg_predict_upper
<int> <int> <int> <int> <dbl> <dbl> <dbl>
1 2 110 6 4 24.8 21.3 28.4Unser Modell macht also eine Vorhersage von etwa 24,8 Meilen pro Gallon mit einem 80 % Vorhersage-Intervall von 21,3 bis 28,4. Das ist nah am tatsächlichen Wert, der in mtcars mit 21,0 angegeben bist.
Yippieh! Das Kommandozeilen-Tool ist damit fertig für den Einsatz als KI-Lösung in unserem Kubernetes-Cluster! Oder einfach nur als Spielerei für zwischendurch …
Worauf sollten Sie achten, wenn Sie ein Kommandozeilen-Skript entwickeln möchten? Ein paar Tipps, die Ihnen dabei helfen können:
Sina Daba
Marketing Managerin
Sobald wir einen Beitrag in unserem Blog veröffentlichen, schicken wir Ihnen gerne eine E-Mail. Und keine Sorge – wir mögen auch keinen Spam!
„*“ zeigt erforderliche Felder an
AM 09. Juli 2025, 14-15 Uhr
Dashboard-Gestaltung praxisnah erklärt. Jetzt anmelden!
VERGANGENE WEBINARE
Ein Webinar darüber, wie Hassia mit Dashboards abteilungsübergreifend den Umgang mit Daten verändert

 5 Tipps für effizientes Arbeiten in Tableau
5 Tipps für effizientes Arbeiten in TableauSie müssen den Inhalt von reCAPTCHA laden, um das Formular abzuschicken. Bitte beachten Sie, dass dabei Daten mit Drittanbietern ausgetauscht werden.
Mehr InformationenSie sehen gerade einen Platzhalterinhalt von Facebook. Um auf den eigentlichen Inhalt zuzugreifen, klicken Sie auf die Schaltfläche unten. Bitte beachten Sie, dass dabei Daten an Drittanbieter weitergegeben werden.
Mehr InformationenSie müssen den Inhalt von reCAPTCHA laden, um das Formular abzuschicken. Bitte beachten Sie, dass dabei Daten mit Drittanbietern ausgetauscht werden.
Mehr Informationen